情報・システム研究機構
ライフサイエンス統合データベースセンター
小野 浩雅 hono@dbcls.rois.ac.jp
2015年5月21日 AJACS御茶ノ水@東京医科歯科大学
これは統合データベース講習会 AJACS御茶ノ水「遺伝子発現DB・ウェブツールの使い方 基礎編」の資料です。
続いて、講習予定の応用・実践編 があります。
講習会全体のプログラムはこちらです。
本講習は、だれでも自由に使うことができる公共データベースやウェブツールを活用して、研究のさまざまな場面で調べることの多い個々の遺伝子発現データを簡単に調べるための方法と基礎知識について学びます。
また、自ら行なった大規模発現解析の(あるいは公共データベースから取得・解析した)結果として得られた数百〜数千におよぶ遺伝子セットについて、生物学的な解釈をする方法とその結果の考察を実践します。
今回の講習では、コンピュータを使って以下の内容について説明します。
- 研究現場で頻繁に使われるデータベースやツールを知る
- 統合TV
- 個々の遺伝子の発現プロファイルを調べる
- RefEx
- 【実習1】RefExを使って、組織特異的遺伝子を検索する
- RefEx
- 数十~数千の遺伝子群の生物学的解釈
- DAVID
- 【実習2】DAVIDを用いて、発現データの結果を生物学的に解釈する
- DAVID
- みんなで同時にアクセスするとサイトにつながりにくくなることが予想されます。
- 資料を見ながら自力で進められそうな方はどんどん先に、そうでない方は講師と一緒にすすめていきましょう。
- サイトの反応が悪い時はタイミングをずらして実行してみてください。
- 反応が無いからと言って何度もクリックするとますます繋がらなくなってしまいます。おおらかな気持ちで臨みましょう。
- わからないことがあったら挙手にてスタッフにお知らせください。
- 遠慮は無用です(そのための講習会です!)。おいてけぼりは楽しくありません。
- 本講習内容をスムーズに理解するために押さえておくとよい基礎知識
- 非モデル生物のデータをモデル生物のデータに見立てるためのID対応表づくりについて
- 次世代シーケンス解析について
- さらにもっと基礎から次世代シーケンス解析について知りたい方向け
- 生命科学分野の有用なデータベースやツールの使い方を動画で紹介するウェブサイト
-
YouTube版もあります http://www.youtube.com/user/togotv/videos
-
ウェブサイトへのアクセスから結果の見方まで、操作の一挙手一投足がわかります。
- 講義・講習などの参考資料や後輩指導の教材として利用できます。
- 本講習中、本家サイトが繋がらない時は、統合TVのYouTube版を見ればおおよその内容がわかるようになっています。
- 今回の講習に関連する内容の多くは、統合TV の発現制御解析 カテゴリーにあります。
- 過去の講習会の内容はそのほとんどが統合TVに収録されており、いつでもどこでも繰り返し復習できるようになっています。
-
お探しの動画が見つからない or 統合TV未掲載の場合は、統合TV番組リクエストフォームへどうぞ!!
-
統合TVを作ってみたい方、募集中です。
- ヒト、マウス、ラットの遺伝子発現情報リファレンスデータセット
- http://refex.dbcls.jp/
- 4つの異なる実験手法(EST、GeneChip、CAGE、RNA-seq)によって得られた正常組織、初代培養細胞、細胞株における遺伝子発現データを検索、閲覧可能
- 最近新たに、FANTOM5 CAGEデータが追加(ヒト556種、マウス286種)
- 掲載しているデータやオリジナルデータなどの詳細については、RefExについて
- RefExで掲載されているデータはすべて再利用可能
- このツールでできること
- 正常組織における遺伝子発現データを調べる
- 測定手法による遺伝子発現量の差異を比較する
- 組織特異的遺伝子をワンタッチで検索可能
- 遺伝子発現解析などで見出された不詳な遺伝子群の機能および関係性を調べる
####【実習1】RefExを使って、組織特異的遺伝子を検索する
- http://refex.dbcls.jp/ を開きます。
- 画面中央の「組織特異的に発現する遺伝子を見る」の臓器アイコンにカーソルを合わせると、更に詳細な部位のアイコンが出るので、調べたい臓器(例として肝臓)をクリックします。
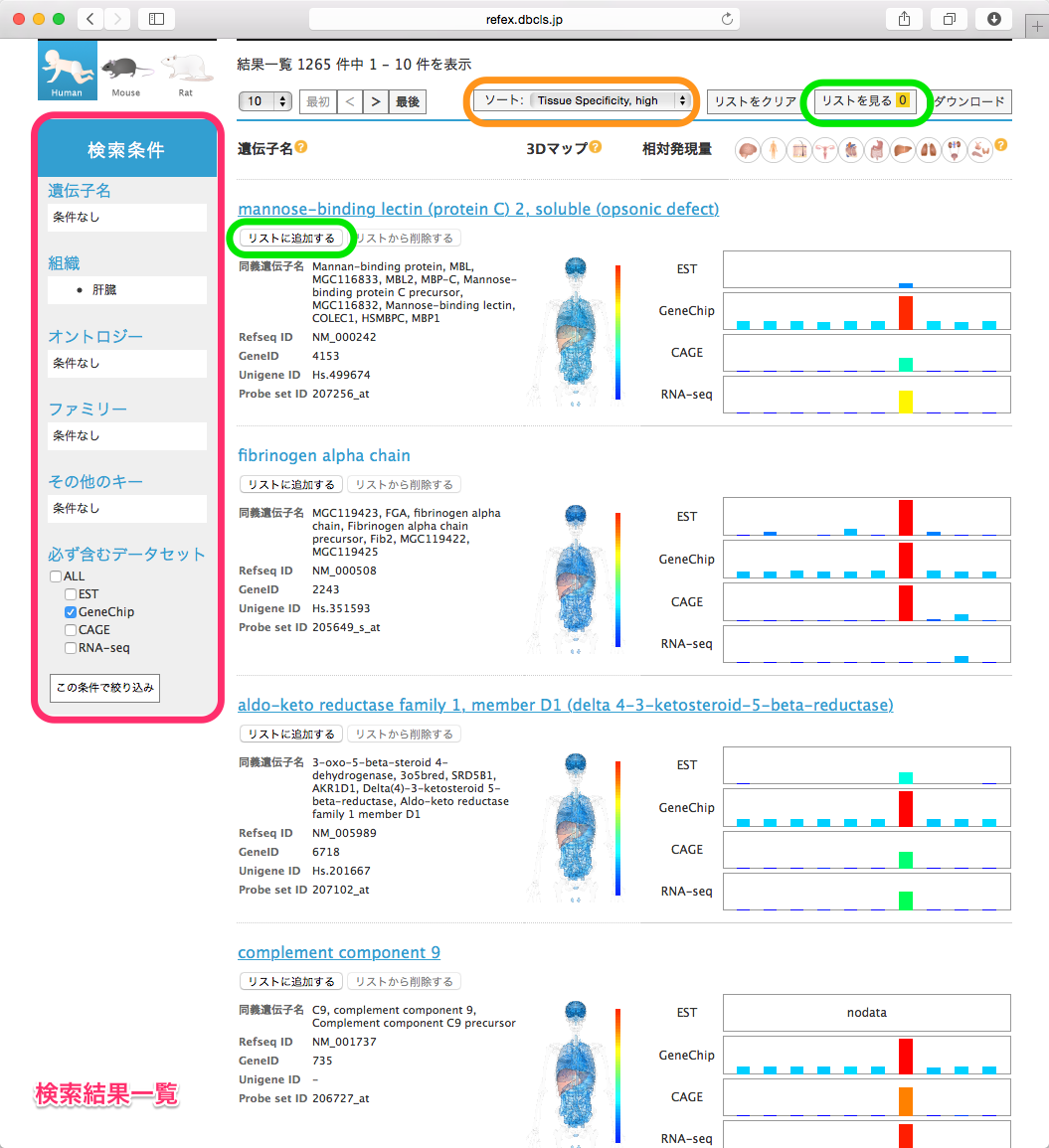
- 検索結果一覧が表示されます。検索結果一覧では、「ソート項目の切り替え」や「絞り込み検索」、「リストへの追加」ができます。(手順11以降で解説します。)
- 各遺伝子の青字の部分(例 fibrinogen alpha chain)をクリックすると詳細情報を閲覧できます。
- 「ヒートマップ on Bodyparts3D」では、表示する部位の切り替え(全身・体幹部・頭部)ができます。「皮膚・骨格筋を表示」もしくは「アニメーション表示」にチェックを入れるとどのように表示されるでしょうか。
- 「組織40分類別データ」では、バーの上にマウスオーバーすると測定部位と発現値が表示されます。
- 「Download」をクリックすると、表示中の遺伝子の組織40分類別の発現データがタブ区切り形式でダウンロードできます。
- 「Probe set ID」のリンク先をクリックすると、どういう情報が参照できるでしょうか。
- 遺伝子オントロジー(GO ID)をクリックすると、そのGO termを持つ他の遺伝子を一括で検索できます。
- 例として、GO:0007596 blood coagulation をクリックしてみましょう。
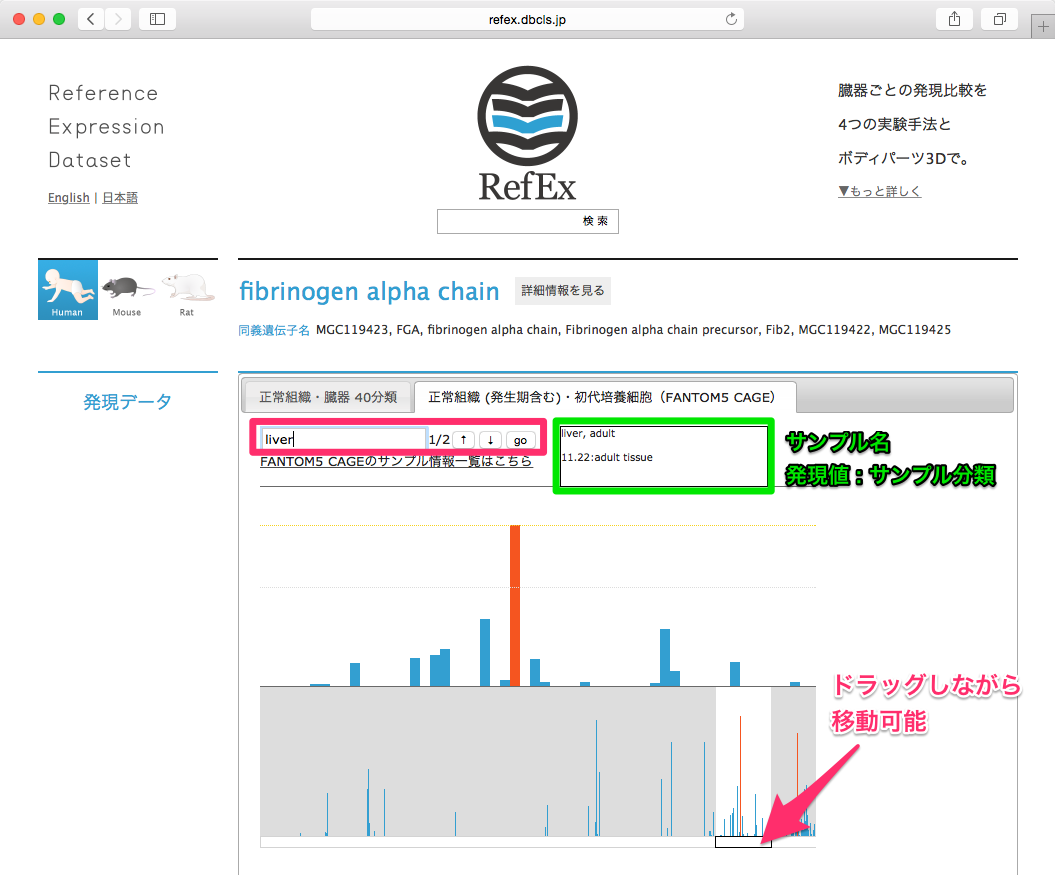
- 右側のFANTOM5 CAGEのタブをクリックすると、FANTOM5 CAGEデータのビューアに切り替わります。
- ビューアは上部が拡大図で、下部が全体表示になっています。
- 検索窓にキーワードを入れるとサンプル名を検索できます。ヒットしたサンプルはオレンジ色で強調されます。
- 右側に、サンプル名と発現値、サンプル分類が表示されます。
- RefEx用に整理したサンプル情報一覧も閲覧可能です。
- 検索結果一覧に戻ります。ソート項目を切り替えて、どのように結果が変わるでしょうか。
- 様々な条件で検索結果を絞り込むことができます。絞り込み検索は左のバーから行えます。
- 遺伝子名に「liver」を含むデータは何件あるでしょうか。
- 「遺伝子名」の下の「条件なし」をクリックして表示されるウインドウに「liver」と入力し、「Include」をクリックし、「この条件で絞り込み」を押します。
- 「遺伝子名」の項目で「Exclude」に「solute」を加えると、検索結果はどう変わるでしょうか。
- 「組織」の項目で、データ元をRNA-seqに変更したり、臓器の指定を追加すると検索結果はどう変わるでしょうか。
- 「必ず含むデータセット」の「ALL」にチェックを入れると、検索結果はどう変わるでしょうか。
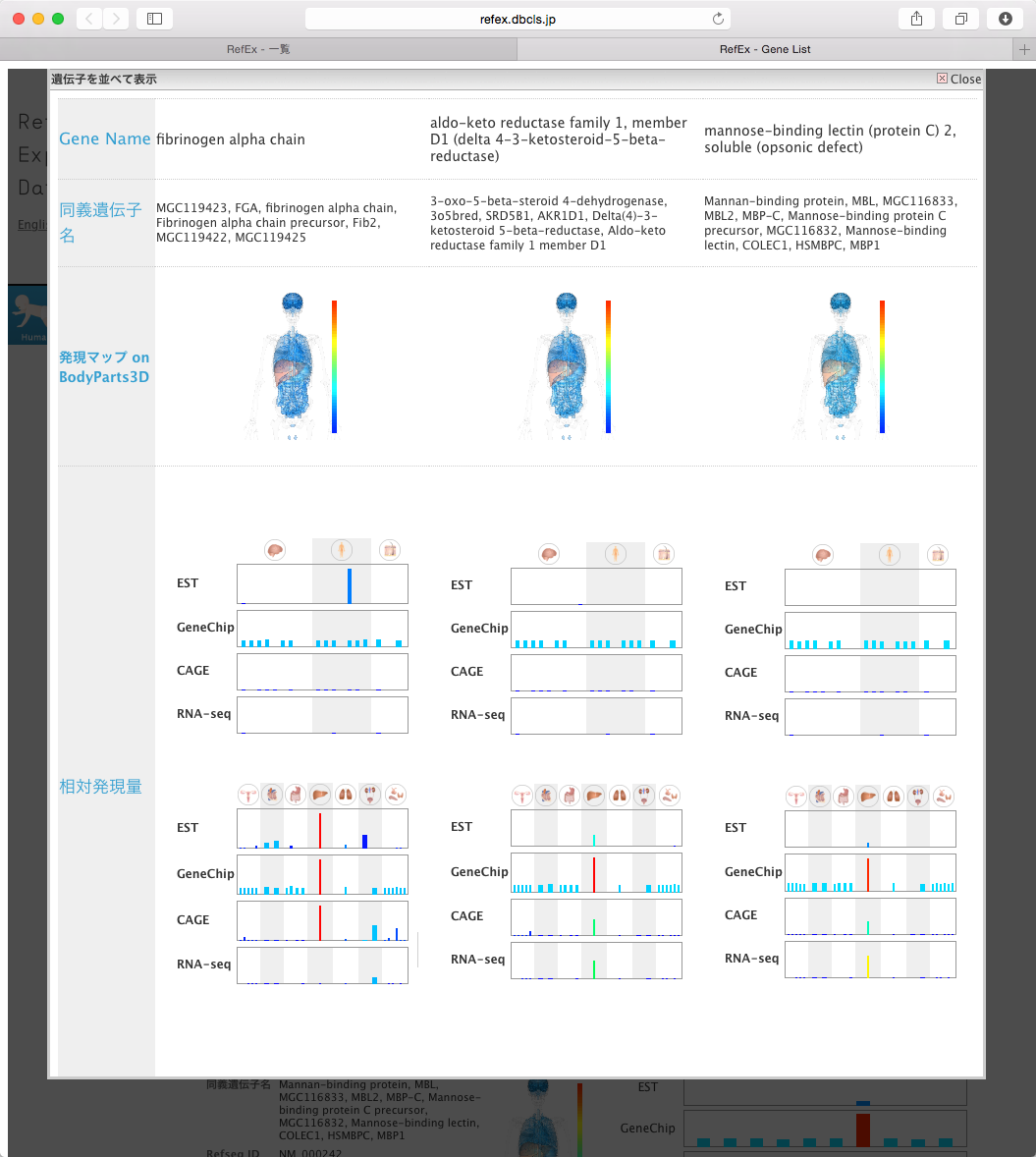
- 個々の遺伝子の詳細情報は、リストに追加することで並列に比較することができます。
- 肝臓特異的遺伝子の検索結果一覧に移動して、3つの遺伝子を「リストに追加」してみましょう。
- 追加した件数は「リストを見る」の横に表示されます。
- 「リストを見る」をクリックするとリストに移動します。
- 『並べて表示する』にチェックを入れて、「遺伝子を並べて表示」をクリックします。
- 並列に比較することで見えてくる「違い」はなんでしょうか。
- 自分の研究テーマに関連する、また興味のある遺伝子について検索してみましょう。
-
ヒト、マウス、ラットのさまざまな組織や細胞(株)における遺伝子発現プロファイルのデータベース
-
BioGPSはAffymetrix社製のマイクロアレイであるGeneChipを用いたさまざまな組織や細胞(株)遺伝子発現プロファイルのデータベース。
-
検索した遺伝子に対して、種々の外部データベースを横断検索することができるだけでなく、それらの設定を保存したり、表示方法を自由にカスタマイズすることができる「Gene annotation portal」。
-
外部データベースには、Wikipedia(Gene Wiki)、著名な試薬会社の検索窓へのリンク集、pathway、Nature系DB、モデル生物DB、文献DBなど多種多様
-
マウスのエキソンアレイのデータから遺伝子のスプライシングバリアント(Splicing variant)の発現状況も調べることが可能。最近ではCircadian関係のデータも。
-
さらに最近のアップデートで、NCBI Gene Expression Omnibus (GEO)中から選抜されたデータセットに切り替えて発現状況を調べることが可能に。
####【実習(skip)】BioGPSを使ってある遺伝子の発現プロファイルを調べる
- http://biogps.org/を開きます。 2.骨格筋の分化決定遺伝子であるMyogenic differentiation 1(MyoD)の発現プロファイルを調べてみましょう。中央の検索窓に「myod」と入力し、「search」を押します。
- 表示された検索結果の中から「ID 4654」をクリックします。
- 最初はヒトのマイクロアレイデータが表示されます。
- 画面左側の"Current Gene List"は右上の<<アイコンをクリックすると非表示にできます。非表示にすることで画面を広く使うことができます。
- ページ内のウインドウは通常のウインドウと同じようにドラッグによる移動やサイズの変更などを行うことができます。 歯車マークのメニューから"Open in browser" を選択すると、新しいタブで表示できます。
- "Search" と書かれた窓に単語(組織名など)を入力すると、その単語の含まれた部分が赤くハイライト表示されます。今回は "Muscle" と入力してみます。
- "Zoom" のバーを用いることで、グラフの表示範囲を調整することが出来ます。
- 発現量を示すバーをクリックすると発現強度の値が表示されます。
- マイクロアレイデータ右上の"Species: Hs"をクリックするとマウスやラットを選択できるので、"M. musculus (Mouse)"をクリックしてマウスのデータを表示できます。
- MyoDはどの組織、細胞で強く発現しているでしょうか?
- 場合によっては"Probeset"のプルダウンメニューから複数の項目を選択できる場合があります。これはどのようなケースが考えられるでしょうか?
- "Static Image" をクリックすると、ズームや検索機能などのついていない、画像だけのグラフで表示されます。低スペックなマシンでは、こちらの方が軽快に動作するでしょう。
- "Correlation"タブをクリックして検索すると、発現パターンが似ている他の遺伝子を検索できますが、どのような遺伝子が出てくるでしょうか?
- "Downloads" をクリックすると現在表示している遺伝子の発現データを CSV 形式でダウンロードできます。
- "Dataset"の右にある'change"をクリックすると、デフォルトで用意されているデータセットやNCBI GEO中のデータセットを検索でき、それらのデータに表示を切り替えることができます。"Species: Hs"に切り替えてから、"change"をクリックしたあと、"Default Datasets"から"Barcode on normal tissues (262 samples)"を選択します。どのようにデータが変わったでしょうか。
- さらに"Search"からキーワード検索で、GEOのデータを検索してみましょう。"C2C12"と検索するとどのようなデータが選択できるでしょうか。
- 右上の「default rayout」をクリックすると、検索した遺伝子に関して種々の外部データベースを横断検索できますが、どのようなデータが閲覧できるのか調べてみましょう。
- 左上の「Search」タグをクリックして検索画面にもどり、自分の興味ある遺伝子について同様に検索してみましょう。 すぐに自分の興味ある遺伝子が浮かばない場合は、著名なiPS細胞を作るために必要な4因子(Oct3/4・Sox2・Klf4・c-Myc)がどの組織で発現しているか、またデータを切り替えて検索してみましょう。
- 【余談】 BioGPSのiPhoneアプリが無料で公開されていますので、「あの遺伝子はどの組織で発現してるのかな?」とふと調べたいときにお手持ちのiPhoneで遺伝子発現を調べられます。
- マイクロアレイデータの生物学的な解釈
- マイクロアレイ実験の一般的な目的は、実験条件によって得られた数十~数千の遺伝子群の発現が生物学的にどういう意味を持つかを考えることです。
- 今回は、その方法の一つとして、マイクロアレイの結果にGene Ontologyの用語を付与することで、生物学的な解釈を行います。
-
サンプルデータとして、NCBI GEOから取得した公共の遺伝子発現データを用います。このデータは、ある実験の前後の2群間で有意に発現減少した遺伝子群のリストです。
→ マル秘遺伝子リスト (右クリックして「新しいタブで開く」もしくは「名前を付けてリンク先を保存」してください。)
-
このデータは、どのような実験から得られたデータなのか、どのように解釈できるのかをDAVIDを使って考察してみましょう!
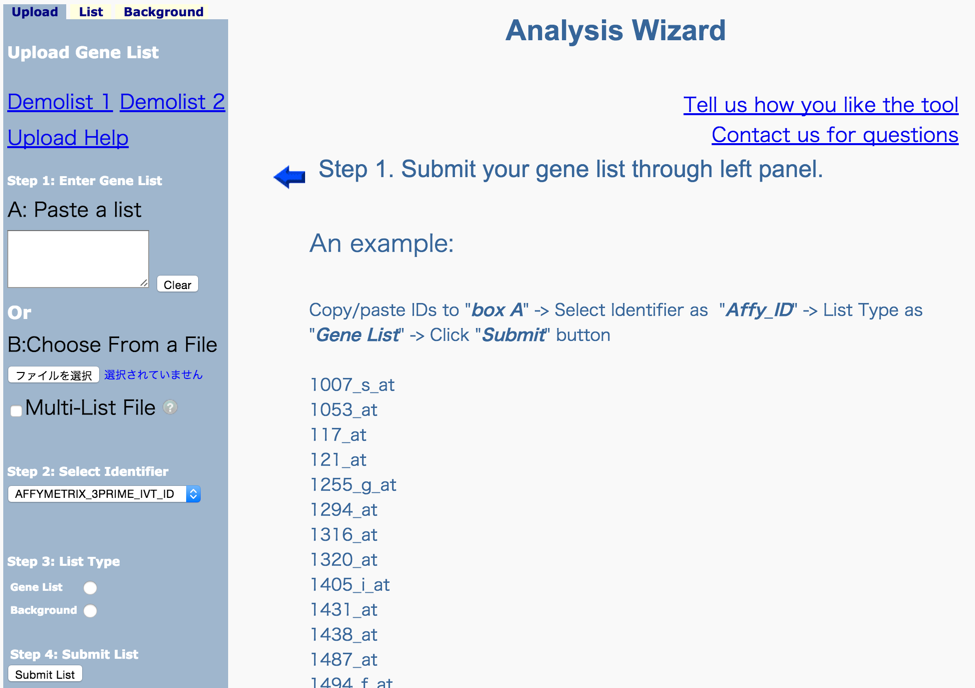
####【実習2】DAVIDを用いて、発現データの結果を生物学的に解釈する
- 上部メニューの「Start Analysis」をクリックします。
- 画面左側バーで、probe IDリストをコピペ or ファイルを指定します。
- リストのIDの種類タイプを選択します。 … 今回は、「AFFY_ID」と「Gene List」
- Submit List をクリックするとリストが読み込まれます。
- アップロードしたリストは、左側バーの「List Manager」で「Uploaded List_1」として保存されています。削除やrenameもできます。
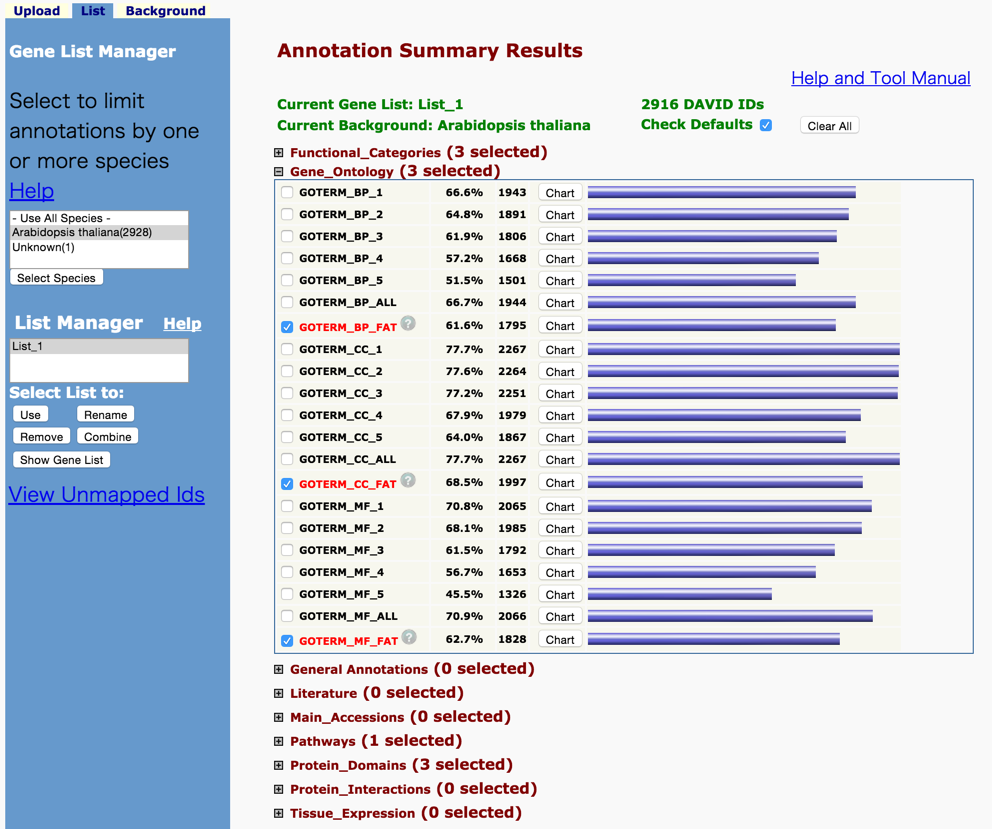
- 解析を続けます。真ん中の「Functional Annotation Tool」をクリックします。
- 「Gene Ontology」をクリックすると、Gene Ontologyを用いた解析の細かいメニューが表示されます。
- 今回は、GOTERM_BP_FAT (BP = Biological Process)に注目します。その右の「Chart」をクリックすると結果がポップアップされます。
- タイトル行をクリックするとソートできます。
- さらに、GOTERM_CC_FAT や GOTERM_MF_FAT を見て、上位にリストされたGOTermにどのような共通点・相違点があるでしょうか。
-
Pathways > KEGG_PATHWAY や Tissue Expression > UP_TISSUE なども見てみましょう。
-
DAVIDで得られた結果を踏まえ、「ある実験」とはどのような実験であったか考察してみましょう。
- マル秘遺伝子リストは「ある実験の前後の2群間で有意に発現減少した遺伝子群のリスト」
- 生物種はArabidopsis thaliana (シロイヌナズナ)