OpenBioblitz est un projet pour une application online/offline pour acquérir de nouvelles données d'observations biologiques lors d'un événement naturaliste comme un BioBlitz.
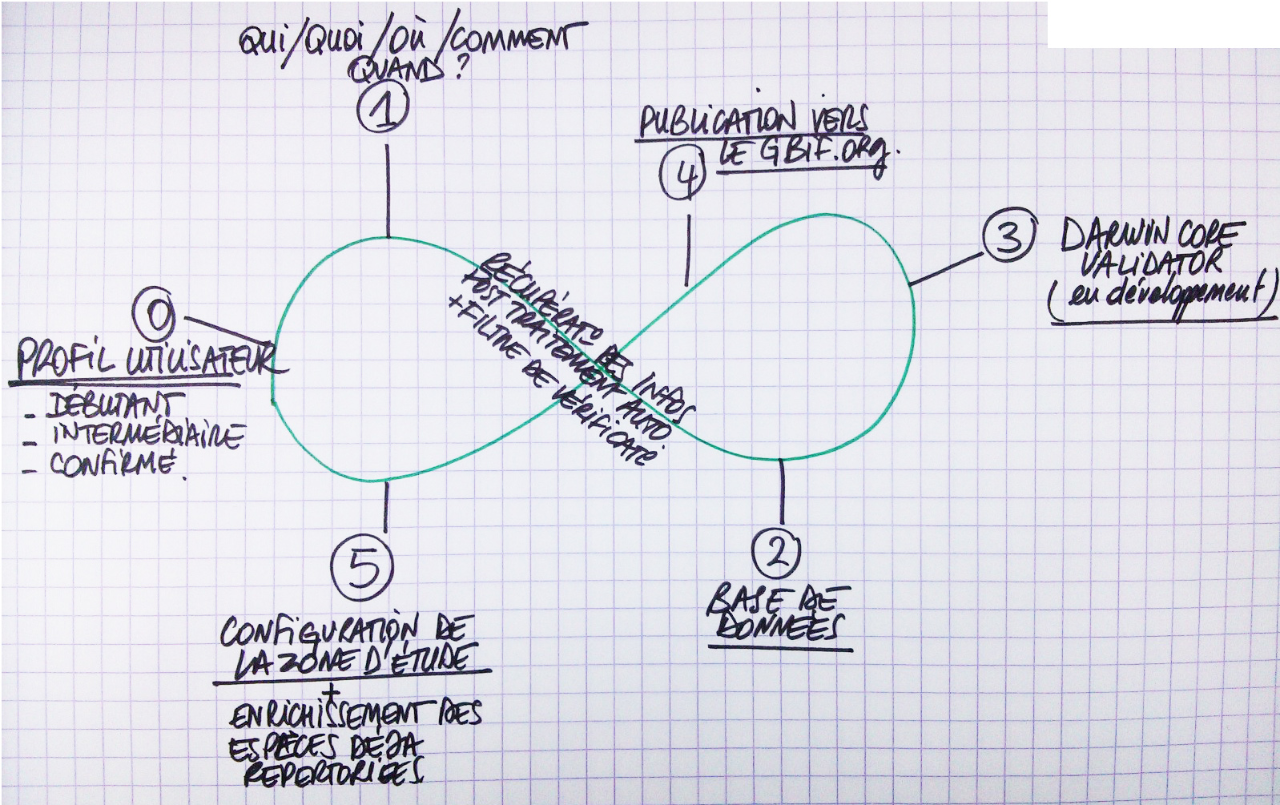
- Traduction automatique en Darwin Core
- Intégration des informations manquantes via des sites OpenSource (OpenTree of Life, Open Street Map, Catalogue of Life, etc.)
- Exportation vers une Base de Données
- Filtrage avec l'outil DarwinCore Validator du GBIF
- Exportation et publications des données vers le GBIF
- Configuration/Paramétrage des espèces déjà présentes sur les lieux via GBIF et/ou l'INPN pour une restriction et optimisation des ajouts d’informations présentes et manquantes
- 3 niveaux de récupération d'information: débutant / intermédiaire / confirmé
Deux interfaces opérationnelles en mode offline (utlisateurs et DarwinCore) échangent avec une API reliée à une Base de Données en ligne. Avant d'être exportées vers le GBIF, les données sont filtrées par l'outil du GBIF DarwinCore Validator.
L'utilisateur choisi son profil : débutant, intermédiare ou confirmé. Les 3 niveaux doivent être très simples pour ne pas perdre l'utilisateur.
<iframe style="width:1200px;height:800px;border:0px solid black" src="https://framindmap.org/c/maps/320935/embed?zoom=1"> </iframe>L'utilisateur rempli un formulaire qui répond au questions Qui / Quoi / Où / Quand / Comment.
Les champs du formulaire sont décris dans cette mind map.
TODO : Vulgariser le DarwinCore selon le type de profil, voir exemple mind map débutant
Connexion avec les API existantes :
- Geographique : OpenStreetMap / GBIF
- Taxonomique : Catalogue of Life
- Phylogénétique : Open Tree of Life
- Images : Encyclopedia Of Life / WikiSpecies
Validation avec DarwinCore Validator.
GBIF.org > occurrence > location (aller voir sur location)
Les choix technologiques ne sont pas arrêtés, chaque proposition est bienvenue.
Choix technologiques :
- Langage : Ruby
- Base de données : PostgreSQL avec extensions (PostGIS, OpenFTS) et éventuellement autres outils (Elasticsearch / Alglia / HStore / Redis ?) à définir.
Todo :
- Décrire les points d'entrées / routes.
Choix technologiques :
- React + Redux
Todo :
- A t-on réellement besoin de plusieurs clients ?
Source : tiré de "Carnet de terrain électronique" et modifié
<iframe style="width:1200px;height:800px;border:0px" src="https://docs.google.com/spreadsheets/d/1tC9VsJVBiUESoh7YoTw2YBSfmn7ER17RecpbAtuqAFE/edit?usp=sharing/"></iframe>TODO: finir de remplir le tableau.
OpenBioBlitz est un projet initié et développé lors d'un hackathon organisé en 32 jours les 4-5 mars 2017 à Rennes (Bretagne, France) dans le cadre de l'événement national la Nuit du Code Citoyen .
Présentation sur les données de biodiversité en modèle ouvert par Olivier Norvez sous licence CC-BY
Todo:
- contacter des institutions (associations naturalistes, Muséums (UMS PatriNat), ou autres projets d'applications naturalistes
OpenBioBlitz est un projet sous licence CC-BY