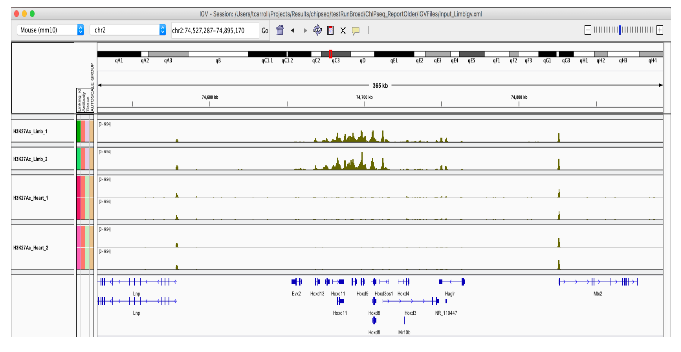
今天我们将继续回顾我们在上一次研究的 Myc ChIPseq。
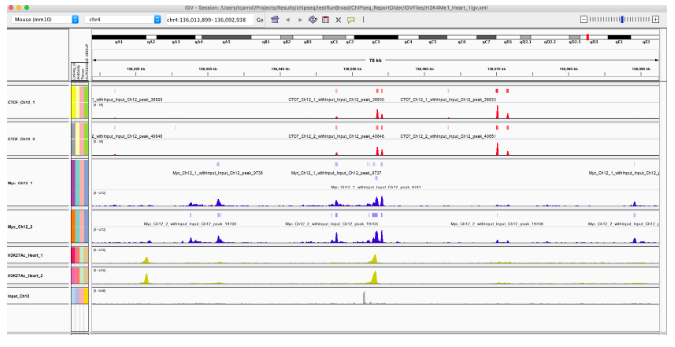
这包括用于 MEL 和 Ch12 细胞系的 Myc ChIPseq。
我按照上一节中概述的处理步骤提供了来自 MACS2 的峰值调用。
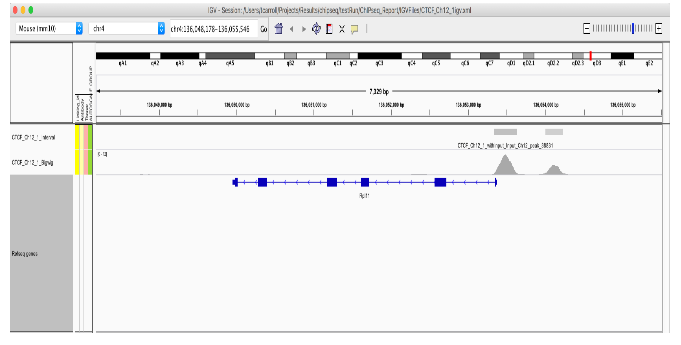
MEL 和 Ch12 细胞系中 Myc 的峰值调用在下面
ChIPseq 的一个共同目标是表征全基因组转录因子结合位点或表观遗传状态。
转录因子结合位点和表观遗传事件的存在通常在其假定目标基因的背景下进一步分析,以表征转录因子和表观遗传事件的功能和/或生物学作用。
随着 ENCODE 对转录因子结合位点或表观遗传状态的大规模映射的发布以及用于高通量测序的多重技术的出现,重复 ChIPseq 实验已成为普遍做法,以便对已识别的表观遗传事件具有更高的信心。
除了表观遗传事件的全基因组表征外,ChIPseq 已越来越多地用于识别条件和/或细胞系之间表观遗传事件的变化。
我们一直致力于处理和表征 Mel 细胞系中的 Myc ChIPseq 复制品。
在本次会议中,我们将研究如何在 Mel 细胞系中定义一组高置信度/可重复的 Myc 峰,以及如何识别 Mel 和 Ch12 细胞系之间独特或常见的 Myc 结合事件。